�ȥåץڡ��� | �ץ��ե����� | �������� | �������� | ���� | ���Υ˥塼�� | ������ | ����¾ | ��°��˾������ | English
�������� †
�ڥץ�����������ʬ������ †
- Takashi Tajimi, Naoki Wakui, Keisuke Yanagisawa, Yasushi Yoshikawa, Masahito Ohue, Yutaka Akiyama: Computational prediction of plasma protein binding of cyclic peptides from small molecule experimental data using sparse modeling techniques, BMC Bioinformatics, 19(Suppl 19): 527, 2018. doi: 10.1186/s12859-018-2529-z
[ PubMed | Journal Website ]
Ligand-based Virtual Screening †
ɸŪ���Ф��벽��ʪ�γ�������եǡ����Ȥ���̤�Τβ���ʪ�γ�����ͽ¬���뵡���ؽ���ˡ�椷�Ƥ��ޤ���
Learning-to-Lank †
- Masahito Ohue, Shogo D. Suzuki, Yutaka Akiyama: Learning-to-rank technique based on ignoring meaningless ranking orders between compounds, Journal of Molecular Graphics and Modelling. doi: 10.1016/j.jmgm.2019.07.009 (in press)
[ PubMed | Journal Website | github ]
- Shogo D. Suzuki, Masahito Ohue, Yutaka Akiyama: PKRank: A novel learning-to-rank method for ligand-based virtual screening using pairwise kernel and RankSVM, Artificial Life and Robotics, 23(2): 205-212, 2018. doi: 10.1007/s10015-017-0416-8
[ Journal Website ]
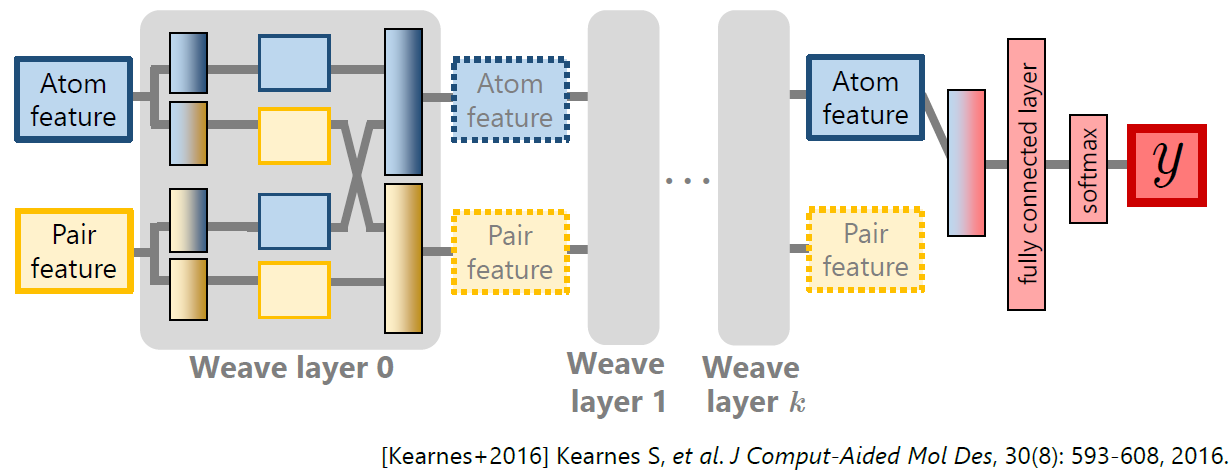
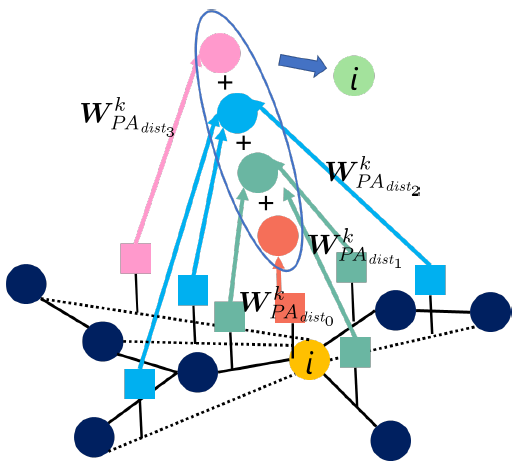
Graph Convolutional Deep Neural Network †
- Masahito Ohue, Ryota Ii, Keisuke Yanagisawa, Yutaka Akiyama: Molecular activity prediction using graph convolutional deep neural network considering distance on a molecular graph, In Proceedings of the 2019 International Conference on Parallel and Distributed Processing Techniques & Applications (PDPTA'19), 122-128, Las Vegas, NV, USA, July 29, 2019.
[ Paper | arXiv | Slide ]
Drug-Target Interactionͽ¬ †
����ѥ��������ޤΣ����ͥåȥ����ͽ¬���뿷����ˡ��ȯ���Ƥ��ޤ���
NRLMF�� †
Betaʬ�ۤ����Ѥ�����������˵��§���ե������ƥ��å��������ʬ��ˡ��ȯ���ޤ�����
- Tomohiro Ban, Masahito Ohue, Yutaka Akiyama: NRLMF��: Bata-distribution-rescored Neighborhood Regularized Logistic Matrix Factorization for Improving Performance of Drug–Target Interaction Prediction, Biochemistry and Biophysics Reports, 18:100615, 2019. doi: 10.1016/j.bbrep.2019.01.008
[ PubMed | Journal Website | github ]
BO-DTI †
�٥�����Ŭ�� (GP-MIˡ) �ˤ�ä�DTIͽ¬�γؽ��ˤ�������֤���10�ܹ�®�����ޤ�����
- Tomohiro Ban, Masahito Ohue, Yutaka Akiyama: Efficient Hyperparameter Optimization by Using Bayesian Optimization for Drug-Target Interaction Prediction, In Proceedings of the 7th IEEE International Conference on Computational Advances in Bio and Medical Sciences (ICCABS 2017), 8 pages, Orlando, FL, USA, October 19-21, 2017. doi: 10.1109/ICCABS.2017.8114299
[ Download* | IEEE Xplore | Slide ]
��*2017 IEEE. Personal use of this material is permitted. Permission from IEEE must be obtained for all other uses, in any current or future media, including reprinting/republishing this material for advertising or promotional purposes, creating new collective works, for resale or redistribution to servers or lists, or reuse of any copyrighted component of this work in other works.
http://www.github.com/akiyamalab/BO-DTI
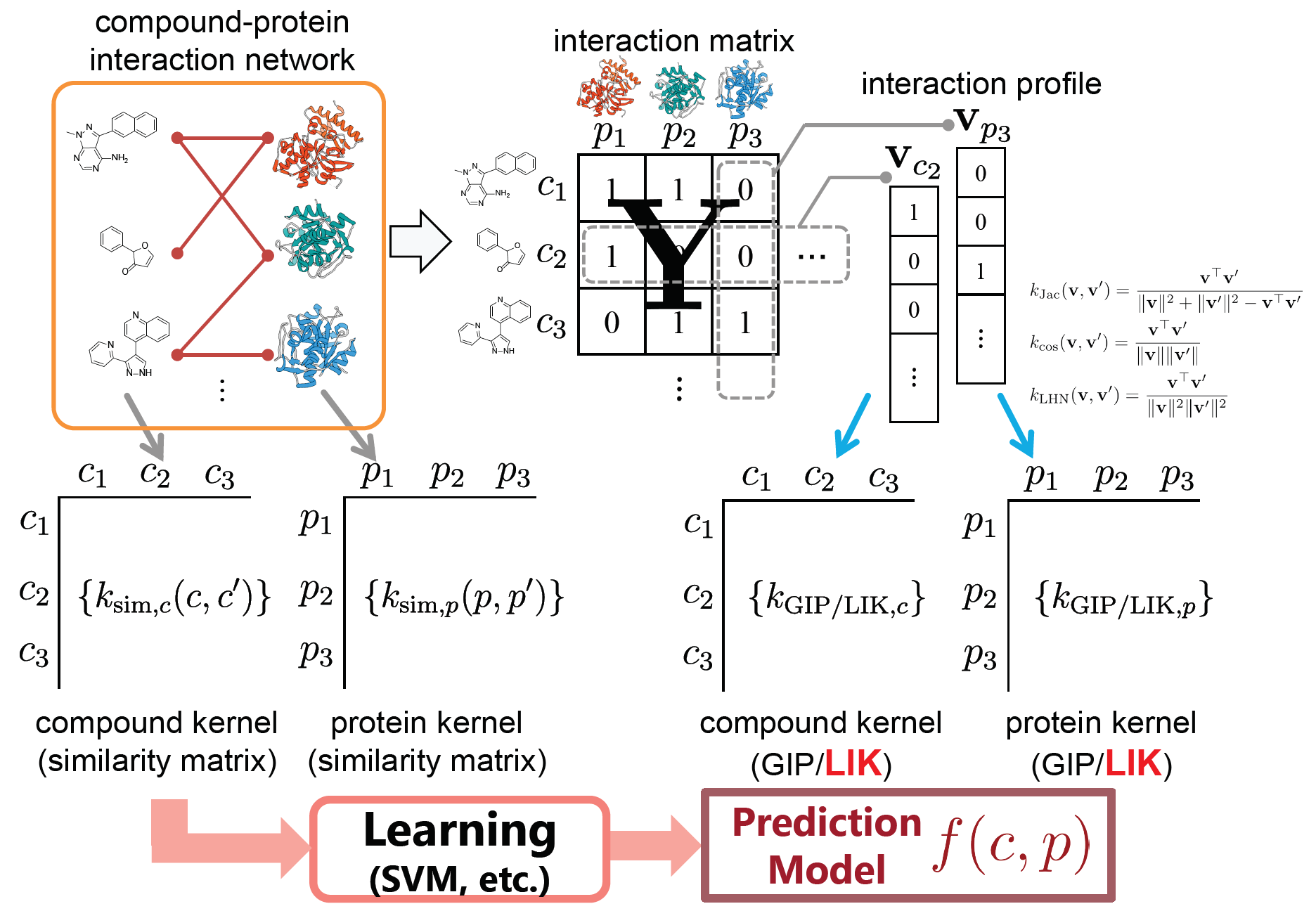
Link Indicator Kernel †
���ɸ���Ѥ����ޥ���ץ륫���ͥ�ˡ����Ƥ��ޤ�����
- Masahito Ohue, Takuro Yamazaki, Tomohiro Ban, Yutaka Akiyama: "Link Mining for Kernel-based Compound-Protein Interaction Predictions Using a Chemogenomics Approach", In Proceedings of the Thirteenth International Conference On Intelligent Computing (ICIC2017) (Lecture Notes in Computer Science), 10362, 549-558, Liverpool,UK August 7-10, 2017.
[ Paper | arXiv | Slide ]
Spresso †
����ѥ�����3������¤�˴�Ť�����®�ʥץ졦������˥����ޤ���
http://www.bi.cs.titech.ac.jp/spresso
- Keisuke Yanagisawa, Shunta Komine, Shogo D. Suzuki, Masahito Ohue, Takashi Ishida, Yutaka Akiyama: Spresso: An ultrafast compound pre-screening method based on compound decomposition, Bioinformatics, 33(23): 3836-3843, 2017. doi: 10.1093/bioinformatics/btx178
[ PubMed | Journal Website ]
- Keisuke Yanagisawa, Shunta Komine, Rikuto Kubota, Masahito Ohue, Yutaka Akiyama: Optimization of memory use of fragment extension-based protein-ligand docking with an original fast minimum cost flow algorithm, Computational Biology and Chemistry, 74: 399-406, 2018. doi: 10.1016/j.compbiolchem.2018.03.013 (In Proc. APBC2018)
[ PubMed | Journal Website | slide ]
MEGADOCK-Web †
- Takanori Hayashi, Yuri Matsuzaki, Keisuke Yanagisawa, Masahito Ohue*, Yutaka Akiyama*: MEGADOCK-Web: an integrated database of high-throughput structure-based protein-protein interaction predictions, BMC Bioinformatics, 19(Suppl 4): 62, 2018. doi: 10.1186/s12859-018-2073-x (In Proc. APBC2018)
[ PubMed | Journal Website | slide ]
- ������, �ӹ���, ������: ����ѥ�������ߺ��Ѥ�ʣ���ι�¤��ͽ¬��̤��Ǥ��륦���֥����ȡ�MEGADOCK-Web��, �¸����, 2019ǯ6���, Vol.37, No.9, pp. 1469-1474, 2019/5/20.
[ ���ڼ� ]
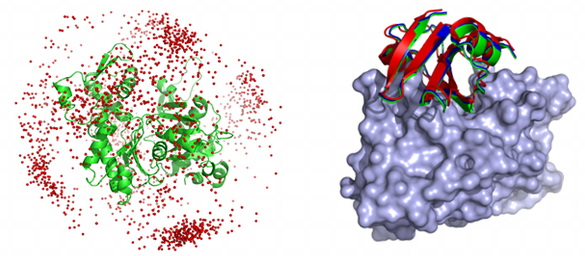
MEGADOCK †

����ѥ�����Ω�ι�¤��������Ѥ��륿��ѥ�������ߺ���ͽ¬�����ƥ�Ǥ���
FFT-base�Υɥå����르�ꥺ���Ƨ�����ʤ���⡤��������ǥ�β��ɤ䥹��å����ʤɤˤ�ä�
��®�ʷ���¸����Ƥ��ޤ���
�ܤ����Ϥ����顧���եȥ�����
NVIDIA Application Catalog�ˤ�Ǻܤ���Ƥ��ޤ���https://www.nvidia.com/en-us/data-center/gpu-accelerated-applications/catalog/
����ѥ����֥ɥå���ͽ¬ †
����ѥ�����Ω�ι�¤���顤�����λ���������Ʊ�Τ��������ʷ����̱��������ˤ�ɽ���Ų٤���ߺ��Ѥʤɤ���Ϥ��ơ�ʣ���Τη����®��ͽ¬������ˡ�椷�Ƥ��ޤ���
- Masahito Ohue†, Yuri Matsuzaki†, Nobuyuki Uchikoga, Takashi Ishida, Yutaka Akiyama: "MEGADOCK: An all-to-all protein-protein interaction prediction system using tertiary structure data", Protein and Peptide Letters, 21(8): 766-778, 2014.
[ PubMed | BenthamScience ]
�������ܥ����륹������ǥ�γ�ȯ †
rPSC�ȸƤФ���ȼ��η�����������������ǥ��١����Ȥ���������ߺ��Ѥ�æ���¼�ͳ���ͥ륮�����ǥ벽��������®�˷���ǽ�ʥ�������ǥ����Ƥ��Ƥ��ޤ���
- Masahito Ohue, Yuri Matsuzaki, Takashi Ishida, Yutaka Akiyama: "Improvement of the Protein-Protein Docking Prediction by Introducing a Simple Hydrophobic Interaction Model: an Application to Interaction Pathway Analysis", In Proceedings of The 7th IAPR International Conference on Pattern Recognition in Bioinformatics (PRIB2012), Lecture Notes in Bioinformatics 7632, 178-187, Springer Heidelberg, 2012.
[ SpringerLink | Slide | Streaming(e-Bio) ]
High Performance Computing (HPC) †
��������ʥ����ѡ�����ԥ塼���ˤ����Ѥ�������ѥ����ɥå���ͽ¬����Ԥ����եȥ�������ȯ���Ƥ��ޤ���
- Masahito Ohue†, Takehiro Shimoda†, Shuji Suzuki, Yuri Matsuzaki, Takashi Ishida, Yutaka Akiyama: "MEGADOCK 4.0: an ultra–high-performance protein–protein docking software for heterogeneous supercomputers", Bioinformatics, 30(22): 3281-3283, 2014.
[ PubMed | Oxford ]
- Yuri Matsuzaki, Nobuyuki Uchikoga, Masahito Ohue, Takehiro Shimoda, Toshiyuki Sato, Takashi Ishida, Yutaka Akiyama: "MEGADOCK 3.0: A high-performance protein-protein interaction prediction software using hybrid parallel computing for petascale supercomputing environments", Source Code for Biology and Medicine, 8(1): 18, 2013.
[ PubMed | BioMedCentral ]
����ѥ�������ߺ��ѥͥåȥ��ͽ¬ †
����ѥ����֤���ߺ��Ѥϡ�����Ǥι���ȿ�����˦�⳰�Ǥξ�����ã�δ��ܤȤʤꡤ��̿��ư�κ�����٤��Ƥ��ޤ����桹�ϥ���ѥ�����Ω�ι�¤�˴�Ť��ɥå������顤��ߺ��Ѥ륿��ѥ����ڥ����®��ͽ¬������ˡ�椷�Ƥ��ޤ���
- Masahito Ohue†, Yuri Matsuzaki†, Nobuyuki Uchikoga, Takashi Ishida, Yutaka Akiyama: "MEGADOCK: An all-to-all protein-protein interaction prediction system using tertiary structure data", Protein and Peptide Letters, 21(8): 766-778, 2014.
[ PubMed | BenthamScience ]
- ������, ����ͳ��, ����͵��, ��ƣ��Ƿ, ������: ��MEGADOCK��Ω�ι�¤���������Ū����ѥ�������ߺ���ͽ¬�Ȥ��Υ����ƥ���ʪ�ؤؤα��ѡ�, ��������ز���ʸ�� ������ǥ벽�ȱ���, 3(3): 91-106, 2010.
[ IPSJ Digital Library ]
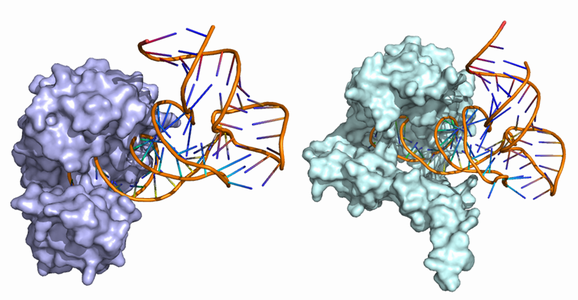
����ѥ���-RNA����ߺ���ͽ¬ †
�ҥȥ��Υ���ˤ�non-coding RNA����1500�Ĥۤɥ����ɤ���Ƥ���Ȥ����Ƥ��ꡤ����ѥ���Ʊ�Τ����Ǥʤ���¿����RNA������ѥ�������ߺ��Ѥ��Ƥ��ޤ����桹���ȼ��Υ���ѥ�������ߺ���ͽ¬�μ�ˡ����Ѥ���������������ѥ���-RNA����ߺ���ͽ¬�μ�ˡ�椷�Ƥ��ޤ���
- Masahito Ohue, Yuri Matsuzaki, Yutaka Akiyama: "Docking-calculation-based Method for Predicting Protein-RNA Interactions",
Genome Informatics, 25(1): 25-39, Aug 3 2011.
[ PubMed | J-stage ]
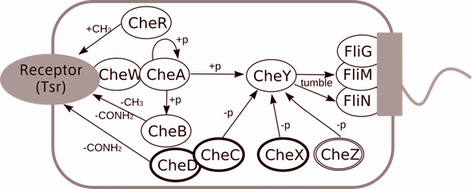
�ٶ��������ѥ����������Ϥؤα��� †
��������λɷ�˱������Ʊ�ư�������������������ȸƤӤޤ���
�㤨�С���IJ�ݤϵ�����֤ˤʤ�ȡ��٤��Ӥΰ����Ҥ�ȯ������
���ܤȤʤ�ʪ���Τ��¿�����ذ�ư���벽���������������ˤ��ޤ���
����������¸����Ƥ���Τϡ��ɷ�˱����ƷϤ��������֤��Ѥ������˦��Υ����ʥ���ã�ϤǤ���
�����ξ��֤��Τ����������֤��Ѥ��Ĥ��б�����Ȥ�����ư�ϡ�
�ؽ��Ȥ�����ǰ�ˤ�Ĥʤ������Ѷ�̣�������ݤǤ���
�ޤ����������Ϥˤ�ä����椵���٤��ӥ⡼�����ϡ�
�����ƥ��ͥ륮����Ψ���ɤ�ʬ�ҥ⡼�����Ȥ��Ƥ����ܤ��졤
����ʬ��ؤα��Ѹ�����Τ��Ƥ��ޤ���
�ܸ���Ǥϡ����ηϤδ��Τ�PPI�������פ��������MEGADOCK��ɾ����Ԥ��ȤȤ�ˡ�
���ηϤ�����˴ؤ��̤�Τ���ߺ��Ѥθ��Ф��ߤޤ�����
- Yuri Matsuzaki†, Masahito Ohue†, Nobuyuki Uchikoga, Yutaka Akiyama: "Protein-protein interaction network prediction by using rigid-body docking tools: application to bacterial chemotaxis", Protein and Peptide Letters, 21(8): 790-798, 2014.
[ PubMed | BenthamScience ]
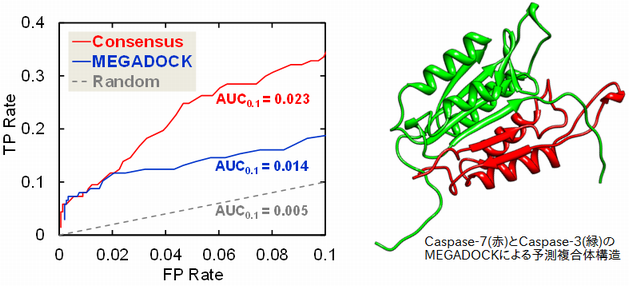
�ҥȥ��ݥȡ������ѥ����������Ϥؤα��� †
���ݥȡ������Ȥϡ����Τξ��֤��ɹ����ݤĤ�����Ѷ�Ū�˰�����������롤
���椵�줿��˦�μ��������Ǥ���
���ݥȡ������θ����˵���������¤Ȥ��ƴ�伫���ȱּ��������ꡤ
���ݥȡ����������ä˵��������ΤȤ��Ƥ�AIDS�����������������졤
�����μ��¤˴ؤ�������������åȤȤ��ơ����ݥȡ������ѥ���������Υ���ѥ����������ܤ���Ƥ��ޤ���
���θ���Ǥϡ�MEGADOCK�ˤ�äƥ��ݥȡ������˴�Ϣ���륿��ѥ�������ߺ��ѥͥåȥ����ͽ¬����ȤȤ�ˡ�����ʣ���ι�¤��ƥ�ץ졼�Ⱦ���Ȥ������Ѥ����ˡ���Ȥ߹�碌�뤳�Ȥ�Ŭ��Ψ�ι⤤ͽ¬���Ԥ��뤳�Ȥ��ޤ�����
- Masahito Ohue, Yuri Matsuzaki, Takehiro Shimoda, Takashi Ishida, Yutaka Akiyama: "Highly Precise Protein-Protein Interaction Prediction Based on Consensus Between Template-Based and de Novo Docking Methods", BMC Proceedings, 7(Suppl 7): S6, 2013.
[ PubMed | BioMedCentral ]
|
![[PukiWiki] [PukiWiki]](image/pukiwiki.png)